Laboratório de Morte em Unicelulares
Inserido no CIIB
(Centro Interdisciplinar de Investigação Bioquímica)
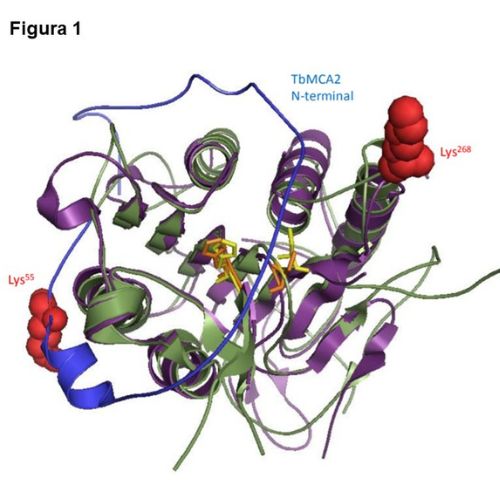
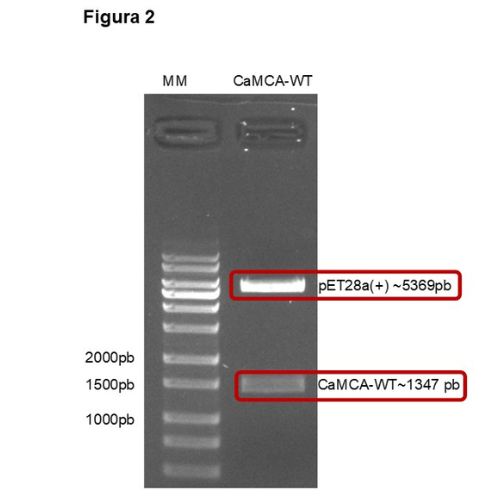
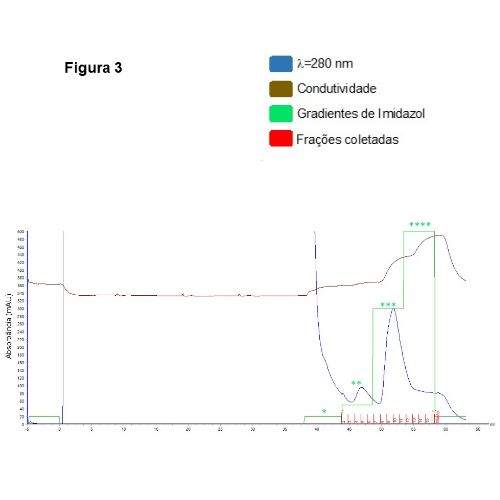
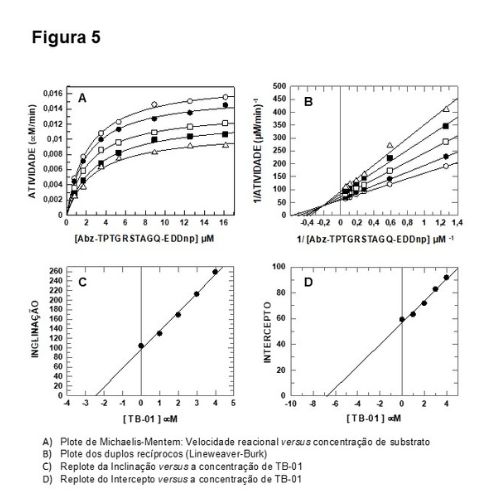
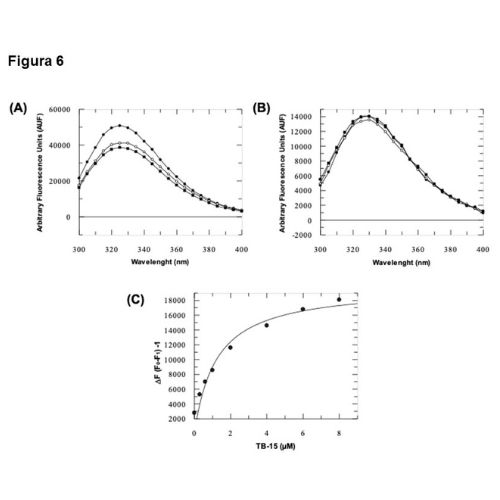
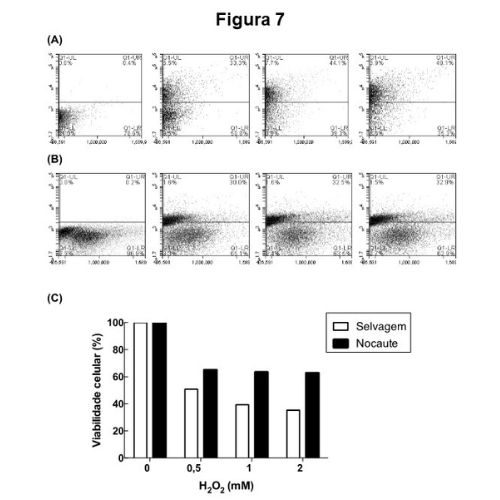
na UMC, nosso grupo estudo o efeito in vivo e in vitro das Metacaspases, que são enzimas proteolíticas envolvidas na morte celular programada (apoptose) em organismos unicelulares como leveduras, protozoários e fungos. Realizamos clonagem e mutações sítio dirigidas para caracterização química, físico-química e estrutural destas enzimas. Também avaliamos o efeito in vivo da metacaspase de Saccharomyces cerevisiae, através de técnicas de imunofluorescência, citometria de fluxo, entre outras utilizando um modelo nocauteado desta levedura para o gene da metacaspase.